Obesity is a chronic disease caused by both environmental and genetic factors. Epidemiological studies have documented that increased energy intake and sedentary lifestyle, as well as a genetic contribution, are forces behind the obesity epidemic. Knowledge about the interaction between genetic and environmental components can facilitate the choice of the most effective and specific measures for the prevention of obesity. The aim of this study was to assess the association between the FTO, AKT1, and AKTIP genes and childhood obesity and insulin resistance.
MethodsThis was a case–control study in which SNPs in the FTO (rs99396096), AKT1, and AKTIP genes were genotyped in groups of controls and obese/overweight children. The study included 195 obese/overweight children and 153 control subjects.
ResultsAs expected, the obese/overweight group subjects had higher body mass index, higher fasting glucose, HOMA‐IR index, total cholesterol, low‐density lipoprotein, and triglycerides. However, no significant differences were observed in genes polymorphisms genotype or allele frequencies.
ConclusionThe present results suggest that AKT1, FTO, and AKTIP polymorphisms were not associated with obesity/overweight in Brazilians children. Future studies on the genetics of obesity in Brazilian children and their environment interactions are needed.
A obesidade é uma doença crônica sustentada por fatores ambientais e genéticos. Estudos epidemiológicos documentaram que maior ingestão de energia e um estilo de vida sedentário, bem como a contribuição genética, são forças por trás da epidemia de obesidade. O conhecimento sobre a interação entre os componentes genéticos e ambientais pode facilitar a escolha das medidas mais efetivas e específicas para a prevenção da obesidade. O objetivo deste estudo foi avaliar a relação entre os genes associado à massa de gordura e à obesidade (FTO), homólogo 1 do oncogene viral v‐akt de timoma murino (AKT1) e de ligação AKT1 (AKTIP) e a obesidade infantil e a resistência à insulina.
MétodosEstudo de caso‐controle no qual os polimorfismos de nucleotídeo simples (SNPs) nos genes FTO (rs99396096), AKT1 e AKTIP foram genotipados em grupos de controle e de crianças obesas/acima do peso. Foram recrutadas 195 crianças obesas/acima do peso e 153 indivíduos controle.
ResultadosComo esperado, os indivíduos do grupo obeso/acima do peso apresentaram maior índice de massa corporal, maior glicemia de jejum, índice do modelo de avaliação de homeostase (HOMA‐IR), colesterol total, lipoproteína de baixa densidade e triglicerídeos. Contudo, não encontramos diferenças significativas no genótipo de polimorfismos gênicos ou nas frequências alélicas.
ConclusãoNossos resultados sugerem que os polimorfismos AKT1, FTO e AKTIP não estavam associados à obesidade/sobrepeso em crianças brasileiras. São necessários estudos futuros sobre a genética da obesidade em crianças brasileiras e suas interações ambientais.
A obesidade infantil é um problema de saúde pública em todo o mundo. Nas últimas décadas, as taxas de sobrepeso e obesidade entre crianças aumentaram em grande parte tanto nos países desenvolvidos quanto nos mais pobres.1 A obesidade é resultado de fatores ambientais que interagem com um histórico poligênico. A herdabilidade varia de 40% a 70%.2 Uma análise de metarregressão atual mostrou que a herdabilidade era maior em crianças do que em adultos.3 Estudos de longo prazo mostraram que a obesidade infantil leva ao agrupamento de componentes da síndrome metabólica (SM),4 que inclui obesidade abdominal, dislipidemia, resistência à insulina, diabetes tipo II e hipertensão.5 É bem sabido que crianças acima do peso e obesas têm um risco maior de se tornar obesas na vida adulta.6
Os estudos de associação genômica ampla (GWAS) identificaram que o gene associado à massa de gordura e à obesidade (FTO) está associado à adiposidade humana.7 O gene FTO está relacionado ao risco de obesidade, principalmente o SNP rs9939609, que posteriormente foi confirmado por outros estudos independentes em diferentes populações humanas.8–11 Devido à relação próxima entre a diabetes e a obesidade, outro gene interessante é o homólogo 1 do oncogene viral v‐akt de timoma murino (AKT1), que, acredita‐se, media muitos efeitos metabólicos, mitogênicos e antiapoptóticos da insulina, fator de crescimento semelhante à insulina tipo 1 (IGF‐1), IL‐3 e outros fatores de crescimento e citocinas.12–14 Ademais, o AKT também estimula a captação de glicose e a síntese de glicogênio,14 bem como a síntese proteica.15 Vários estudos correlacionam a resistência à insulina a comprometimentos no caminho do AKT e, em algumas doenças, essas alterações podem ser de origem genética. A proteína de ligação AKT1 (AKTIP ou Ft1), localizada próximo ao FTO no mesmo locus de risco de GWAS de obesidade no cromossomo 16 (16q12.2),16 parece ser outro alvo para investigação. A AKTIP, como ligante direta do AKT, modula a integração e sinaliza caminhos.17
O equilíbrio energético é influenciado por uma série de variáveis, como dieta, estruturas sociais, fatores metabólicos, estilo de vida sedentário moderno, abundância de alimentos baratos com alto nível calórico e genética. Provavelmente, a obesidade comum é o resultado de um ambiente adverso obesogênico, que trabalha em um genótipo suscetível.4 Até hoje, a evidência dos possíveis benefícios clínicos de estudos genéticos para doenças complexas comuns tem sido limitada. Mesmo assim, a busca por fatores genéticos envolvidos na obesidade é um desafio e pode fornecer dados extras para responder a essa questão complexa. Por meio dessas observações, desenvolvemos este estudo com o objetivo de investigar a associação estatística de polimorfismos nos genes FTO, AKT1 e AKTIP com obesidade infantil em crianças brasileiras.
MétodosDados clínicosOs pais e os filhos foram informados sobre a natureza e o objetivo deste estudo e assinaram um consentimento por escrito. Todas as crianças foram submetidas a um exame físico completo. A estatura foi medida com um estadiômetro de parede, ao passo que o peso corporal foi obtido com uma balança digital. O índice de massa corporal (IMC) foi calculado com as medições de peso e estatura de acordo com a equação IMC=peso (kg)/estatura2 (cm). Os pontos de corte do IMC adotados foram aqueles estabelecidos pela OMS.18 Sobrepeso e obesidade foram definidos como um IMC superior a desvio padrão de+1 e+2, respectivamente.19 A pressão arterial foi medida pelo menos duas vezes e a presença de hipertensão foi definida como PAS e/ou PAD acima do 95° percentil.20 Nenhuma das crianças estava sob o uso de medicação.
O perfil lipídico e a glicemia foram determinados em soro e plasma, respectivamente, com kits comerciais de métodos enzimáticos de rotina (Labtest Diagnóstica, S.A., Brasil). Os níveis de insulina foram medidos com um kit (Genese Produtos Diagnósticos, São Paulo, Brasil). A resistência à insulina foi calculada pelo índice do modelo de avaliação de homeostase (HOMA‐IR), conforme descrito por Wallace et al.21 Até hoje, poucos estudos definiram os níveis de corte para HOMA‐IR em indivíduos pré‐púberes e púberes. Com base nisso, o HOMA‐IR foi testado como uma variável contínua e foi controlado pela situação do IMC dos indivíduos.
O comitê de ética local aprovou este estudo sob o protocolo CAAE 06400000180‐7.
Pacientes acima do peso e obesosNossa amostra incluiu 195 obesos/acima do peso com média de 11 anos (± 3,9) (104 meninas), IMC de 26,7 (± 4,5). Todos foram recrutados do Ambulatório de Endocrinologia do Instituto da Criança e do Adolescente em Juiz de Fora e do Ambulatório de Endocrinologia Pediátrico da Fundação Instituto Mineiro de Estudo e Pesquisa em Nefrologia (Imepen) em Juiz de Fora e residiam em áreas urbanas.22,23
Grupo de controleAs pessoas no grupo de controle foram cuidadosamente escolhidas da comunidade local e consistiam em 153 pessoas magras e normotensas com média de 11,9 anos (± 3,2) (91 meninas), IMC de 17,9 (± 2,3). Todos forneceram um consentimento informado por escrito e o Comitê de Ética da Universidade aprovou os protocolos.
GenotipagemO DNA genômico foi isolado com o método de teor elevado de sal após a coleta de sangue periférico em tubos a vácuo.24 As sondas selecionadas corresponderam aos seguintes SNPs: FTO: rs9939609; AKTIP: rs9302648, rs7189819; AKT1: rs2494738, rs3730358, rs10149779. Para melhorar o poder estatístico, escolhemos apenas SNPs com menor frequência do alelo >0,2 em pessoas brancas e iorubás na base de dados HapMap. A genotipagem foi feita com proteína C reativa (PCR) em tempo real com um 7.500 Real‐Time PCR System (Applied Biosystems Inc., CA, EUA), na modalidade de discriminação alélica. Os protocolos de PCR foram feitos de acordo com as instruções do fabricante do TaqMan® Genotyping Master Mix (Applied Biosystems, CA, EUA).
Análise estatísticaAs frequências alélicas, haplotípicas e genotípicas foram comparadas entre grupos com o teste X2 e o software Unphased (Unphased, v.3.0.13, Cambridge, Reino Unido).25 O teste de 1.000 permutações (teste posterior) foi feito em cada teste para estimar a relevância global de todas as análises e validar os valores de expectativa‐maximização. O software Haploview (Haploview, v 4.1, Broad Institute, MA, EUA) foi usado para avaliar as matrizes de desequilíbrio de ligação (DL) entre cada SNP para examinar a estrutura em blocos do DL e o equilíbrio de Hardy‐Weinberg (corte de 0,05).26 Após a correção de Bonferroni, o nível de significância foi estabelecido em 0,0083 para as comparações do grupo do genótipo x. As características clínicas do grupo de obesos/acima do peso foram comparadas com aquelas das crianças e adolescentes controle por análise de variância (Anova) unidirecional, seguida do teste de Tukey post hoc. Para verificar a detecção de interações entre genes associadas a doenças humanas complexas, usamos a redução multifatorial da dimensionalidade (MDR) e a redução multifatorial da dimensionalidade – teste de desequilíbrio (MDR‐PDT).27
Pelo menos 10% das amostras foram regenotipadas aleatoriamente para fins de controle de qualidade, o que minimizou erros de genotipagem. Foi obtida uma concordância de 100% entre os resultados de ambos os testes.
Uma análise secundária testou se os SNPs estudados em nossa pesquisa estavam linearmente associados ao HOMA‐IR e ao IMC. Usamos um modelo de regressão linear para testar a relação de cada variável preditora independente (SNP) com cada resultado metabólico (HOMA‐IR ou IMC). Várias regressões lineares eram usadas se mais de uma variável preditora mostrasse relação com cada resultado.
ResultadosSeis SNPs foram genotipados em 348 indivíduos. Todos os SNPs estavam em equilíbrio de Hardy‐Weinberg e apresentaram menor frequência do alelo (MAF) acima de 10%, o que indica uma boa penetração dos alelos na população.
As características clínicas e laboratoriais dos grupos estudados são apresentadas na tabela 1. Como esperado, os indivíduos do grupo obeso/acima do peso apresentavam maiores índices de massa corporal. Em comparação com o grupo de controle, o grupo obeso/acima do peso mostrou maior glicemia de jejum, índice de HOMA‐IR, colesterol total, lipoproteína de baixa densidade e triglicerídeos (p=<0,01) (tabela 1).
Características demográficas dos participantes do estudo
| Controle n=153 (%) | Obesidade n=195 (%) | |
|---|---|---|
| Sexo feminino | 91 (59,47) | 104 (53,33) |
| Idade | 11,9 ±3,2 | 11 ±3,9 |
| IMC | 17,9 ±2,3 | 26,7 ±4,5 |
| PAS | 105,3 ±12,4 | 118,7 ±15,1 |
| PAD | 65,2 ±9,7 | 73,8 ±9,7 |
| Hipertensão | 5 (3,2) | 61 (31,2) |
| Glicose (mg/dL) | 80,8 ±10,9 | 86 (±8,8) |
| HOMA‐IR | 2,17 ±2,21 | 5,68 ±3,28 |
| Colesterol total | 142 ±43 | 145 ±35 |
| LDL | 60,5 ±34 | 84,8 ±35 |
| HDL | 44 ±9 | 39 ±10 |
| Triglicerídeos (mg/dL) | 74,3 ±27 | 88,2 ±41 |
HDL, lipoproteína de alta densidade (mg/dL); HOMA‐IR, modelo de avaliação da homeostase – resistência à insulina; IMC, índice de massa corporal (kg/m2); LDL, lipoproteína de baixa densidade (mg/dL); PAS, pressão arterial sistólica (mmHg); PAD, pressão arterial diastólica (mmHg).
Valores apresentados como média±DP.
Sexo e hipertensão são números de amostras (%).
A tabela 2 apresenta a distribuição de genótipos e alelos nos grupos. Na análise de alelos, o AKT1 rs10149779 apresentou uma associação sem risco com o alelo ancestral G (p=0,2; RC=0,70; X2=4,40; gl=1) entre o grupo obeso/acima do peso e o de controle após o teste de 1.000 permutações (teste posterior). O mesmo polimorfismo também não apresentou associação genotípica G/G (p=0,09; RC=0,61; X2=6,29; gl=2) em comparação a indivíduos eutróficos. O genótipo T/T para o marcador AKTIP rs9302648 também não apresentou associação (p=0,09; RC=1,03; X2=7,15; gl=2) após o teste de 1.000 permutações (teste posterior). Nenhuma associação foi encontrada nos outros marcadores (tabela 2).
Genótipo e frequências de alelos dos seis SNPs marcados dos polimorfismos FTO/AKT1/AKTIP nas amostras
| Obesidade n=195 (%) | Controle n=153 (%) | RC | 90% Baixo 95% Alto | X2 | gl | Valor de p (1.000 permutações) | |
|---|---|---|---|---|---|---|---|
| AKT1 | |||||||
| rs2494738 | |||||||
| G/G | 150 (76,92) | 126 (82,25) | 1 (ref) | 1,13 | 2 | 0,56 | |
| A/G | 30 (15,38) | 18 (11,76) | 1,40 | 0,73‐2,63 | |||
| A/A | 1 (0,5) | 1 (0,65) | 0,84 | 0,05‐13,57 | |||
| Ga | 330 (84,62) | 270 (88,24) | 1 (ref) | 0,83 | 1 | 0,36 | |
| A | 32 (8,2) | 20 (6,54) | 1,30 | 0,73‐2,34 | |||
| rs3730358 | |||||||
| G/G | 116 (59,49) | 90 (58,82) | 1 (ref) | 2,31 | 2 | 0,31 | |
| A/G | 54 (27,69) | 48 (31,37) | 0,87 | 0,54‐1,40 | |||
| A/A | 11 (5,64) | 4 (2,61) | 2,13 | 0,65‐6,23 | |||
| Ga | 286 (73,33) | 228 (74,51) | 1 (ref) | 0,15 | 1 | 0,68 | |
| A | 76 (19,49) | 56 (18,3) | 1,08 | 0,73‐1,59 | |||
| rs10149779 | |||||||
| G/G | 94 (48,21) | 55 (35,95) | 1 (ref) | 6,29 | 2 | 0,04 (0,09) | |
| G/A | 73 (37,44) | 76 (49,68) | 0,56 | 0,35 ‐ 0,89 | |||
| A/A | 18 (9,23) | 17 (11,11) | 0,61 | 0,29 ‐1,30 | |||
| Ga | 261 (37,83) | 186 (60,78) | 1 (ref) | 4,40 | 1 | 0,03 (0,20) | |
| A | 109 (27,95) | 110 (35,95) | 0,70 | 0,51‐0,97 | |||
| AKTIP | |||||||
| rs9302648 | |||||||
| T/T | 76 (38,97) | 46 (30,07) | 1 (ref) | 7,15 | 2 | 0,02 (0,09) | |
| G/T | 76 (38,97) | 82 (53,59) | 0,56 | 0,34‐0,90 | |||
| G/G | 36 (18,46) | 21 (13,73) | 1,03 | 0,54‐1,98 | |||
| Ta | 228 (58,46) | 174 (56,86) | 1 (ref) | 0,34 | 1 | 0,55 | |
| G | 148 (37,95) | 124 (40,52) | 0,91 | 0,66‐1,24 | |||
| rs7189819 | |||||||
| C/C | 67 (34,36) | 61 (39,87) | 1 (ref) | 0,95 | 2 | 0,61 | |
| C/T | 84 (43,08) | 65 (42,48) | 1,17 | 0,73‐1,89 | |||
| T/T | 34 (17,44) | 23 (15,03) | 1,34 | 0,71‐2,53 | |||
| Ca | 218 (55,9) | 187 (61,11) | 1 (ref) | 1,01 | 1 | 0,31 | |
| T | 152 (38,97) | 111 (36,27) | 1,17 | 0,85 ‐1,60 | |||
| FTO | |||||||
| rs9939609 | rs9939609 | ||||||
| A/A | 44 (22,56) | 34 (22,22) | 1 (ref) | 4,42 | 2 | 0,10 | |
| T/A | 45 (23,08) | 71 (46,41) | 1,14 | 0,66‐1,96 | |||
| T/T | 39 (20,0) | 46 (30,07) | 0,65 | 0,35‐1,21 | |||
| Aa | 193 (49,49) | 139 (45,42) | 1 (ref) | 1,88 | 1 | 0,16 | |
| T | 183 (46,92) | 163 (53,27) | 0,80 | 0,59‐1,09 | |||
gl: grau de liberdade; gl=1 entre 2 alelos; gl=2 entre 3 genótipos; RC, razão de chance; uma associação significativa é indicada em negrito itálico (< 0,05); valor de p ajustado pelo teste de permutação: 0,2 (alelo); valor de p ajustado pelo teste de permutação: 0,09 (genotípico); valor de p ajustado pelo teste de 1.000 permutações (múltiplo teste – sem fase).
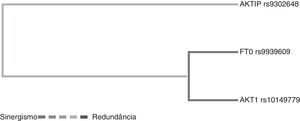
Na análise da interação entre genes por meio da redução multifatorial de dimensionalidade, o dendrograma apresenta uma relação entre os pares de genes. O termo sinergia denota uma situação em que a interação baseada em entropia entre dois SNPs gera mais informações que a correlação entre o par. Ao passo que a redundância é usada nos casos em que a interação entre dois SNPs gera menos informações do que a correlação entre os pares (fig. 1).
Dendrograma da interação gênica de pacientes obesos e controles. As cores da legenda incluem um espectro que representa a transição do sinergismo (preto) para redundância (cinza). Observe as seguintes interações: sinergismo entre o FTO rs9939609 e o AKT1 rs10149779 e redundância entre estes e AKTIP rs9302648.
Quando a análise da resistência à insulina e desses polimorfismos foi concluída, não foi encontrada associação (dados não apresentados).
Embora não tenhamos obtido um valor de p significativo na análise genética do FTO rs9939609, quando a análise gênica foi concluída, foi encontrada uma interação entre o FTO e o AKT1 rs10149779.
A tabela 3 apresenta o resultado dos modelos de regressão linear. Não encontramos associação significativa entre cada SNP e o HOMA‐RI ou o IMC.
Modelo de regressão linear que aborda as contribuições de cada SNP para o HOMA‐IR e o IMC em nossa amostra
| Modelos de regressão linear do HOMA | |||||
|---|---|---|---|---|---|
| Variável preditora | F | p | R2 ajus. | Beta padrão | t |
| AKT1 rs2191738 HEX/FAM A/G | 0,00 | 0,983 | 0,00 | 0,01 | 0,22 |
| AKT1 rs3730358 HEX/FAM A/G | 0,12 | 0,725 | 0,00 | 0,02 | 0,35 |
| AKT1 rs10119779 HEX/FAM A/G | 0,02 | 0,880 | 0,00 | 0,01 | 0,15 |
| FTO rs9939309 HEX/FAM A/T | 0,00 | 0,971 | 0,00 | 0,00 | 0,03 |
| AKTIP rs9302318 HEX/FAM G/T | 0,65 | 0,421 | 0,00 | 0,05 | 0,81 |
| AKTIP rs7189819 HEX/FAM C/T | 0,33 | 0,567 | 0,00 | 0,04 | 0,57 |
| Modelos de regressão linear do IMC | |||||
|---|---|---|---|---|---|
| Variável preditora | F | p | R2 ajus. | Beta padrão | t |
| AKT1 rs2191738 HEX/FAM A/G | 0,01 | 0,919 | 0,00 | 0,00 | 0,10 |
| AKT1 rs3730358 HEX/FAM A/G | 0,06 | 0,802 | 0,00 | −0,01 | −0,25 |
| AKT1 rs10119779 HEX/FAM A/G | 2,23 | 0,137 | 0,00 | −0,08 | −1,49 |
| FTO rs9939309 HEX/FAM A/T | 3,08 | 0,080 | 0,01 | −0,09 | 1,75 |
| AKTIP rs9302318 HEX/FAM G/T | 0,05 | 0,816 | 0,00 | 0,01 | 0,23 |
| AKTIP rs7189819 HEX/FAM C/T | 1,38 | 0,240 | 0,00 | −0,06 | −1,18 |
Beta pad., coeficiente de regressão padronizado; HOMA, índice do modelo de avaliação da homeostase; IMC, índice de massa corporal; R2 ajus., R2 ajustado.
A obesidade é uma doença clínica complexa modulada por muitos fatores genéticos e não genéticos, com diversas interações entre muitos deles. Foram investigados seis SNPs não codificados dos genes AKT1, AKTIP e FTO como possíveis genes da obesidade em crianças brasileiras. Nossos resultados sugerem que os polimorfismos AKT1, FTO e AKTIP não foram associados à obesidade/sobrepeso em crianças brasileiras. Vale observar o fato de que as crianças obesas/acima do peso com o alelo A do FTO rs9939609 não apresentaram risco maior de se tornar obesas nessa população. Esse polimorfismo foi associado ao IMC e à diabetes tipo II em diversas populações, como finlandeses e asiáticos.28–30 Recentemente, os estudos de associação de todo o genoma em crianças brancas31 e chinesas32 reconheceram vários polimorfismos de nucleotídeo simples (SNPs), incluindo o gene FTO, associados ao IMC e à obesidade. Embora diversas variações do gene FTO tenham sido associadas à obesidade em populações com histórico europeu, seus efeitos em outras etnias ainda devem ser estabelecidos. Em um estudo que envolveu 478 crianças negras, não foi encontrada associação entre as variações do gene FTO e o IMC, resultado semelhante a outros estudos com populações negras e gambianas.33 A mistura étnica brasileira pode ser um motivo para a inexistência de associação com um polimorfismo associado à obesidade clássico.
Poucos estudos com relação a variantes do FTO e IMC em crianças e adolescentes latino‐americanos foram publicados até agora. Da Silva et al.34 relataram uma associação significativa entre o genótipo A/A do FTO rs9939609 e aumento de IMC e gordura subcutânea em 348 crianças brasileiras.35 Curiosamente, em outro estudo a respeito de crianças brasileiras, o alelo FTO rs9939609 A foi associado a um aumento de IMC entre crianças com insuficiência de vitamina D, ao passo que nenhum efeito significativo foi encontrado entre aquelas com situação adequada de vitamina D. Esse pode ser um erro do tipo I, mas também pode significar que esse problema é mais complexo do que o esperado e envolveu outros fatores metabólicos.
Até o momento, poucos estudos associaram as variantes no gene AKT1 e a resistência à insulina. Em um estudo recente, um haplótipo que incluiu o primeiro éxon do AKT1 foi associado a maior frequência de síndrome metabólica em indivíduos americanos descendentes de negros e de europeus.36
Quando fizemos a análise gênica (MDR), foi encontrada uma interação entre o FTO rs9939609 e o AKT1 rs10149779. Uma possível razão para essa diferença entre polimorfismo único e os resultados da análise MDR pode ser devido à epistasia, definida como a interação entre dois ou mais genes ou seus produtos proteicos de mRNA, que modulam um fenótipo.37 As análises estatísticas e os modelos animais mostraram que a epistasia é um fenômeno comum na obesidade. Desse modo, os genes da obesidade conferem, isoladamente, pequenos aumentos nas taxas de risco de obesidade. Contudo, podem influenciar o desenvolvimento da obesidade por mecanismos mais complexos, principalmente interações entre genes e entre gene e o ambiente.38
Como o FTO apresentou efeitos maiores sobre o IMC na infância em percentis maiores em alguns estudos, fizemos uma regressão linear.39 Contudo, não foi encontrada associação. Alguns dos achados de associação negativa podem resultar de alguma limitação de nosso estudo, principalmente o tamanho pequeno da amostra total. Ademais, não consideramos fatores ambientais, como os padrões de atividade física, presença de pais acima do peso/obesos, bem como dados relativos à ingestão de alimentos, o que impede uma análise mais detalhada.
A obesidade é uma doença complexa e os mecanismos envolvidos na interação entre genótipo e ambiente ainda precisam ser esclarecidos. Nossos dados podem lançar alguma luz para entender melhor esse problema de saúde complexo e relevante. Deve‐se considerar o viés da publicação existente em estudos com abordagem de genes candidatos. Além disso, muitos estudos sobre a genética da obesidade podem apresentar um poder estatístico inadequado, o que aumenta o risco de associações falso positivas.40
Em resumo, não encontramos evidência de associação entre polimorfismos AKT1, AKTIP e FTO com a obesidade e a resistência à insulina nesta amostra de crianças brasileiras. São muitos os motivos para os resultados contrastantes observados nos estudos, incluindo diferenças no histórico genético das diferentes amostras. Considerando as associações encontradas em outros estudos, acreditamos que os estudos que envolvem um número maior de pessoas de diferentes grupos étnicos terão elucidar a função desses genes com a obesidade. Dessa forma, nosso trabalho enfatiza a importância do cuidado ao analisar estudos de genes candidatos e reforça a necessidade de estudos bem conduzidos para ampliar nosso entendimento dos fatores genéticos e ambientais envolvidos na obesidade e na resistência à insulina.
FinanciamentoCoordenação de Aperfeiçoamento de Pessoa de Nível Superior (Capes), Fundação de Amparo à Pesquisa do Estado de São Paulo (Fapesp), Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq, 573646/2008‐2) e Fundação de Amparo à Pesquisa do Estado de Minas Gerais (Fapemig, CBB‐APQ‐00075‐09 e CDS‐BPD‐00296‐14).
Conflitos de interesseOs autores declaram não haver conflitos de interesses.
Este estudo foi financiado por CAPES, FAPESP, CNPq (FAPEMIG:CBB‐APQ‐00075‐09/CNPq 573646/2008‐2), e FAPEMIG:CDS‐BPD‐00296‐14.
Ambos os autores contribuíram igualmente para este manuscrito.
Como citar este artigo: Pereira PA, Alvim‐Soares AM, Sandrim VC, Lanna CM, Souza‐Costa DC, Belo VA, et al. Lack of association between genetic polymorphism of FTO, AKT1 and AKTIP in childhood overweight and obesity. J Pediatr (Rio J). 2016;92:521–7.