Clinical use of microarray‐based techniques for the analysis of many developmental disorders has emerged during the last decade. Thus, chromosomal microarray has been positioned as a first‐tier test. This study reports the first experience in a Chilean cohort.
MethodsChilean patients with developmental disabilities and congenital anomalies were studied with a high‐density microarray (CytoScan™ HD Array, Affymetrix, Inc., Santa Clara, CA, USA). Patients had previous cytogenetic studies with either a normal result or a poorly characterized anomaly.
ResultsThis study tested 40 patients selected by two or more criteria, including: major congenital anomalies, facial dysmorphism, developmental delay, and intellectual disability. Copy number variants (CNVs) were found in 72.5% of patients, while a pathogenic CNV was found in 25% of patients and a CNV of uncertain clinical significance was found in 2.5% of patients.
ConclusionChromosomal microarray analysis is a useful and powerful tool for diagnosis of developmental diseases, by allowing accurate diagnosis, improving the diagnosis rate, and discovering new etiologies. The higher cost is a limitation for widespread use in this setting.
O uso clínico de técnicas baseadas em microarrays para a análise de transtornos de desenvolvimento tem surgido durante a última década. Assim, o microarray cromossômico tem sido posicionado como um teste de primeiro nível clínico. Relatamos a primeira experiência em uma coorte chilena.
MétodosPacientes chilenos com atraso de desenvolvimento e anomalias congênitas foram estudados com um microarray de alta densidade (CytoScan™ HD Array, Affymetrix, Inc., Santa Clara, CA, EUA). Pacientes tiveram estudos citogenéticos anteriores, ou um resultado normal ou de uma anomalia não bem caracterizada.
ResultadosForam analisados 40 pacientes selecionados por dois ou mais critérios, incluindo: anomalias congênitas maiores, dismorfismo facial, atraso de desenvolvimento e deficiência intelectual. Uma variante do número de cópia (CNV) foi encontrada em 72,5% dos pacientes, enquanto que uma CNV patogênica foi encontrada em 25% dos pacientes e uma CNV de significado clínico incerto foi encontrada em 2,5% dos pacientes.
ConclusõesA análise cromossômica microarray é uma ferramenta útil e poderosa em transtornos de desenvolvimento, permite um diagnóstico preciso, melhora a taxa de diagnóstico e descobre novas etiologias. O custo mais elevado é uma limitação para um uso difundido em nossa realidade.
Anomalias congênitas múltiplas afetam de dois a três indivíduos em cada 100 nascidos vivos e são a principal causa de mortalidade e deficiência infantil.1,2 Apesar de sua maioria ser isolada e de origem multifatorial, pacientes com anomalias múltiplas precisam de uma avaliação para identificar uma causa genética subjacente.
Nos últimos anos, o estudo etiológico de transtornos no desenvolvimento tem sido enriquecido com o uso clínico de técnicas em microarray. Em países desenvolvidos, o cariótipo molecular, ou a análise cromossômica por microarray (CMA), é considerado a técnica de primeira linha para a análise de pacientes com anomalias congênitas múltiplas, atraso de desenvolvimento/deficiência intelectual não sindrômicos e transtornos do espectro autista.3‐7
Em contrapartida, em países em desenvolvimento, como da América Latina, a detecção de anomalias cromossômicas ainda é feita principalmente por técnicas citogenéticas convencionais. O cariótipo com bandamento GTG (banda G pela tripsina e corante giemsa) em linfócitos tem sido amplamente usado principalmente para identificar anomalias cromossômicas com uma resolução maior ou igual a 5‐10 megabases (5‐10 Mb).8‐11 A hibridização in situ por fluorescência (FISH) está disponível para um número limitado de doenças causadas por microdeleções/microduplicações cromossômicas e tem uma resolução de 2‐5 Mb em núcleos metafásicos e entre 50‐150 Kb em núcleos interfásicos.8,9,11‐13 Outras técnicas moleculares foram desenvolvidas para procurar pequenas microdeleções/microduplicações, como a Amplificação de Múltiplas Sondas Dependentes de Ligação (MLPA).14 Em contrapartida a essas técnicas convencionais, a CMA tem maior resolução, que atinge 50 Kb, dez vezes maior do que a resolução do cariótipo convencional.13,15 Essa busca desequilíbrios genéticos (ganhos ou perdas de segmentos cromossômicos) em todo o genoma e tem permitido a identificação de novas síndromes não prontamente detectadas pelos métodos descritos anteriormente.16‐18 A descoberta da variante normal em variantes do número de cópias (CNVs) representa um desafio à interpretação clínica.15
Embora os estudos de diagnóstico para indivíduos com anomalias congênitas ou deficiência intelectual com base na citogenética convencional apresentem rendimento diagnóstico próximo de 3%, a CMA apresenta rendimento de aproximadamente 15‐20%, ou seja, mais de cinco vezes acima do cariótipo com bandamento G,6 o que justifica seu uso como teste de diagnóstico de primeira linha para pacientes com diagnósticos clínicos desconhecidos. Estima‐se que a CMA consiga detectar, individualmente, mais de 99% de todas as anomalias do cariótipo.5
Este relatório apresenta nossa experiência pioneira no uso da CMA em uma coorte de pacientes chilenos com anomalias congênitas múltiplas sem diagnóstico etiológico.
MétodosPacientesForam selecionados 40 pacientes das Clínicas Genéticas do Hospital Padre Hurtado (Santiago, Chile) entre maio de 2012 e novembro de 2012.
Incluímos pacientes que apresentavam no mínimo duas das seguintes características clínicas: anomalias congênitas múltiplas (ACMs), dismorfismo facial (DF), atraso de desenvolvimento (AD) ou deficiência intelectual (DI). Nenhum dos pacientes apresentou uma causa definida da doença.
Dentre todos os pacientes, 36 apresentaram um cariótipo normal, dois apresentaram um pequeno cromossomo marcador supranumerário (sSMC) descaracterizado, um apresentou um cromossomo derivado, um apresentou translocação robertsoniana herdada e um apresentou monossomia do cromossomo X, porém com características adicionais incomuns.
O comitê de ética local aprovou este estudo e o consentimento informado por escrito foi obtido de todos os pacientes e/ou pais.
Processamento das amostrasO DNA genômico foi purificado a partir de células mononucleares do sangue periférico com o Kit Miniprep da AxyPrep para extração de DNA genômico do sangue (Axygen Biosciences, Union City, CA, EUA) de acordo com as instruções do fabricante. O DNA genômico de cada paciente foi hibridizado para o CytoScan™ HD Array (Affymetrix, Inc., Santa Clara, CA, EUA) de acordo com as instruções do fabricante. Essa hibridização genômica comparativa por array de alta densidade personalizada com quase 2,7 milhões de marcadores genéticos inclui 700.000 marcadores de polimorfismo de nucleotídeo simples (SNP) e mais de 1,9 milhão de sondas de oligonucleotídeos não polimórficas para a detecção de CNVs.
Análise de dadosOs dados de array foram analisados com o software da Affymetrix® Chromosome Analysis Software Suite (ChAS) v.1.2.2 (Affymetrix, Inc., Santa Clara, CA, EUA) com base na sequência genômica de referência do UCSC Genome Browser hg19, fevereiro de 2009 (GRCh37/hg19). Analisamos variantes do número de cópias (CNV) de mais de 400 Kb, conforme recomendado.6 Com essa plataforma de maior resolução, conseguimos avaliar uma CNV menor, porém nenhum paciente apresentou anomalias clinicamente relevantes entre 100 e 400 Kb e, dessa forma, mantivemos esse limite para fins deste relatório. As CNVs de mais de 400 Kb foram categorizadas por relevância clínica como CNV de relevância clínica clara (grupo 1), CNV de relevância não clara ou relevância incerta (grupo 2) ou CNV benigna ou polimórfica (grupo 3), com as bases de dados publicamente disponíveis Isca (International Standard Cytogenomic Array),19 DGV (Database of Genomic Variants),20,21 Omim (Online Mendelian Inheritance in Man),22 Decipher (Database of Chromosomal Imbalance and Phenotype in Humans Using Ensembl Resources)23,24 e Ecaruca (European Cytogeneticists Association Register of Unbalanced Chromosome Aberrations).25
ResultadosDos 40 pacientes analisados, 16 (41%) eram do sexo feminino. As idades variavam de um mês a 25 anos, com uma idade média de 4,2 anos. Como selecionado, a vasta maioria de nossos pacientes tem anomalias múltiplas, incluindo transtornos de desenvolvimento estruturais e funcionais. Os detalhes clínicos estão resumidos na tabela 1.
Resumo das características clínicas e dos desequilíbrios genéticos encontrados nessa coorte
CMA, análise cromossômica por microarray; FISH, hibridização in situ por fluorescência; N/A, não aplicável pela idade; NOR, região organizadora do núcleo; sSMC: pequeno cromossomo marcador supranumerário.
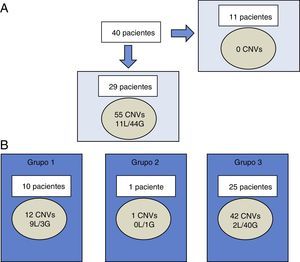
Encontramos 55 CNVs de mais de 400 Kb em 29 de 40 pacientes (72,5%), variação entre 0 e 4 CNVs por paciente. Do total, 11 foram perdas e 44 foram ganhos (fig. 1A). O tamanho dos desequilíbrios cromossômicos variava entre 420,9 Kb e 25,2 Mb. Esse último valor corresponde a um paciente com sSMC detectado por cariótipo.
Caracterização das CNVs maiores do que 400 Kb na coorte chilena.
A, não foram encontradas CNVs em 11 de 40 pacientes. Nos 29 pacientes restantes, foram encontradas 55 CNVs (11 perdas e 44 ganhos). B, cada paciente poderá ter CNV de mais um grupo ao mesmo tempo. As CNVs foram categorizadas por relevância clínica como CNV de relevância clínica clara (grupo 1), CNV de relevância incerta (grupo 2) ou CNV benigna ou polimórfica (grupo 3).
CNVs: variantes do número de cópias; G: ganhos; L: perdas.
Esses 55 CNVs foram classificados em três grupos com base em sua interpretação clínica.
De acordo com nossa classificação (fig. 1B), 21,8% pertenciam ao grupo 1 (relevância clínica clara relacionada ao fenótipo), 1,8% ao grupo 2 (relevância incerta) e 76,4% ao grupo 3 (benigno ou polimórfico). As perdas aconteceram predominantemente no grupo 1, ao passo que os ganhos ocorreram no grupo 3.
Rendimento diagnósticoNo Grupo 1, dois pacientes apresentaram dois desequilíbrios cromossômicos terminais, provavelmente resultantes de translocações crípticas não balanceadas. Sete apresentaram uma microdeleção e um apresentou trissomia mosaico parcial do cromossomo 20 (um dos pacientes com sSMC). Mais detalhes estão resumidos na figura 1.
No Grupo 2, apenas um paciente apresentou uma variação de relevância incerta (VOUS).26 Esse paciente tinha uma triplicação de 2 Mb na posição 9p23 [array 9p23 (9.323.653‐11.359.708) x3], porém, com as informações disponíveis nas bases de dados e na literatura biomédica, não poderíamos descartar nem atribuir um efeito patogênico definitivo.
Não conseguimos encontrar desequilíbrios genéticos verdadeiros no segundo paciente com sSMC e no paciente com cromossomo 16 derivado. No paciente com translocação robertsoniana, não encontramos desequilíbrio genômico que explicasse o fenótipo. Por fim, no paciente com monossomia do cromossomo X, confirmamos a deleção de um cromossomo X, porém não encontramos desequilíbrios genômicos adicionais (fig. 1).
Interpretação clínicaEncontramos uma CNV patogênica não vista anteriormente (n=9) ou não bem caracterizada (n=1) por cariótipo convencional em 10 de 40 pacientes do grupo 1 (25,0%). Além disso, corroboramos uma anomalia citogenética conhecida em um paciente (monossomia do cromossomo X).
Se considerarmos apenas os pacientes com cariótipo normal (35), nossa taxa de diagnósticos aumentou para 28,5%.
Além disso, encontramos uma CNV de relevância clínica incerta ou VOUS (grupo 2) em outro paciente. Esse aguarda testes adicionais e acompanhamento para definir a relevância clínica desse achado. Planejamos estudar ambos os pais por meio de Fish e/ou MLPA.
DiscussãoEste é o primeiro relatório de teste de CMA em uma coorte de pacientes chilenos com deficiências de desenvolvimento, considerando que existem poucos relatórios do uso clínico de microarrays cromossômicos na América Latina, que relatam experiências de casos individuais. Na América do Sul, uma experiência semelhante foi relatada em um grupo de pacientes no Brasil.27,28 Foram analisados 95 pacientes “sindrômicos”’ com cariótipo normal e foi relatado um rendimento diagnóstico de 17%.28
Detectamos mais de 25% de alterações patogênicas em nossa coorte, que estão na mais alta faixa relatada na literatura. Em pacientes com atraso de desenvolvimento/deficiência intelectual sindrômicos e não sindrômicos e cariótipo normal/Fish, a metanálise apresenta um rendimento diagnóstico de 7,8%‐13,8%, variação de 5% a 50%.5,29 Isso é explicado, em grande parte, pela heterogeneidade no modelo dos estudos, principalmente na seleção dos pacientes, testes previamente feitos e plataforma de array usada. Em nosso caso, a alta taxa de diagnóstico pode ser explicada pelo fato de que a coorte estudada é relativamente pequena e apresenta o viés de pacientes muito bem selecionados, muitos dos quais permaneceram sem diagnóstico por muito tempo. Finalmente, usamos uma plataforma de alta densidade.
Com o passar do tempo, diferentes bases de dados públicas on‐line coletaram informações fenotípicas e genômicas de milhares de pacientes anônimos, o que permitiu a compreensão da vasta maioria de CNVs como benignas ou polimórficas, sem análises adicionais. Na verdade, a maioria de nossos achados foi de CNVs polimórficas (Polimorfismos do Número de Cópias [CNP]), corroboradas nas bases de dados citogenômicas e dos dados de nossa própria coorte. Observamos CNVs recorrentes de mais de 400 Kb em metade dos pacientes, principalmente envolvendo os seguintes loci cromossômicos: 10q11.22, 14q32.22, 16p11.2, 17q21.31, Yq11.223 e Yq11.23. Apenas um caso que apresentou uma VOUS exigiu uma análise adicional para determinar a patogenicidade possível. Esse paciente tinha um ano e 11 meses, com DI, atraso de linguagem, deficiência de audição, hérnia inguinal e baixa estatura. Ele apresentou um cariótipo 46,XY e o array mostrou um ganho de 2 Mb no cromossomo 9p23, incluindo a triplicação parcial de um gene: PTPRD. A proteína PTPRD é uma proteína tirosina fosfatase tipo receptor, expressa em certas regiões do cérebro, como o hipocampo, e pode ter uma função no aprendizado e na memória30 e na organização sináptica.31 Nenhum fenótipo foi atribuído à triplicação total desse gene.
Apesar de a CMA ser uma técnica muito forte e confiável, tem limitações: não consegue detectar anomalias genômicas equilibradas, como inversões, translocações recíprocas e robertsonianas. A depender da plataforma usada, o mosaicismo de baixo nível e algumas poliploidias não podem ser detectados. Quando a técnica da CMA é usada como a primeira linha de estudo, descreveu‐se que essa situação pode ser constatada em 0,78% dos casos.5 Por fim, a CMA não fornece informações sobre a posição do rearranjo e a Fish normalmente é usada como método complementar para identificar possíveis rearranjos com implicações para o aconselhamento genético.7,32
Em nossa coorte, descobrimos que três pacientes com anomalias citogenéticas previamente conhecidas resultaram em um cariótipo molecular normal. Como esperado, o paciente portador de uma translocação robertsoniana apresentou um resultado de CGH‐array normal/equilibrado. No caso de um dos dois pacientes com sSMC, a cobertura do array, as sequências genômicas envolvidas e o tamanho do sSMC podem explicar por que esse material genético não foi detectado por esse método.19 Assim, esse pequeno cromossomo bisatélite provavelmente corresponde a sequências altamente repetitivas, típicas de cromossomos acrocêntricos não incluídos no array que foi demonstrado por bandamento da região organizadora do nucléolo (NOR). No paciente com um cromossomo 16 derivado, com um padrão de bandamento anormal e uma morfologia também anormal (mais metacêntrica que o normal), uma inversão pericêntrica é a explicação mais plausível.
Assim, o cariótipo continua sendo mais adequado para avaliar possíveis portadores de rearranjos cromossômicos, casais com abortos recorrentes ou pacientes com um fenótipo característico de aneuploidia. Da mesma forma, a Fish é mais adequada em caso de suspeita elevada de uma síndrome específica de microdeleção.7
A análise cromossômica por microarray é um método altamente preciso, robusto e de alto rendimento. Temos usado um array combinado com base em oligonucleotídeos e um array de SNP, o que cria muitas vantagens, já que o array de SNP melhora de forma significativa a precisão e a sensibilidade da detecção e mosaicismo da CNV e permite igualmente a detecção de variantes neutras de número de cópias.33 No caso do chip usado neste relatório (Cytoscan HD, Affymetrix), a plataforma química e seus algoritmos analisam o oligonucleotídeo e as sondas de SNP de forma independente. Assim, podemos detectar e confirmar cada CNV ao mesmo tempo, o que não exige confirmação adicional.32,33 Nesse caso, em vez de uma confirmação de achado de arrays, a análise FISH permite determinar o tipo de rearranjo.32
O alto custo do exame cria uma limitação no uso difundido do cariótipo molecular. No Chile, o custo da CMA é quatro a sete vezes maior do que o cariótipo e/ou Fish e atualmente não é coberto por plano de saúde. Contudo, para chegar a um diagnóstico precoce em alguns pacientes com anomalias congênitas múltiplas e/ou atraso global de desenvolvimento, é possível evitar exames desnecessários (a “odisseia de diagnóstico”) e permitir foco nos problemas específicos, o que pode ter um bom custo‐benefício em longo prazo.34‐38 Pode ser esperada uma diminuição dos custos com o passar do tempo, o que permitirá seu uso mais difundido. Por fim, deve ser mencionado que existem outras plataformas com resolução mais baixa, porém por um custo relativamente reduzido.
Reconhecendo suas limitações, existe cada vez mais evidência do impacto clínico dessa tecnologia. Em muitos pacientes, um diagnóstico definitivo pode afetar não apenas a informação e o aconselhamento de seu ambiente familiar,39 mas também a vigilância ativa em busca de possíveis complicações, entre outros tipos de intervenções médicas.37,38
Nossos dados mostram a utilidade da CMA, que permite uma capacidade de diagnóstico melhorada com precisão e o aprimoramento da gestão e supervisão da saúde desse grupo de pacientes com necessidades especiais.
FinanciamentoBolsa da Fundação de Saúde Infantil em Birmingham, Alabama.
Conflitos de interesseOs autores declaram não haver conflitos de interesse.
Agradecemos à Fundação de Saúde Infantil em Birmingham, Alabama, por sua bolsa para este trabalho, aos pacientes e parentes participantes e à Dra. Silvia Castillo e à citogeneticista Ana María Fuentes pela análise dos resultados de cariótipo convencional.
Como citar este artigo: Lay‐Son G, Espinoza K, Vial C, Rivera JC, Guzmán ML, Repetto GM. Chromosomal microarrays testing in children with developmental disabilities and congenital anomalies. J Pediatr (Rio J). 2015;91:189–95.