Respiratory syncytial virus is a pathogen frequently involved in nosocomial outbreaks. Although several studies have reported nosocomial outbreaks in neonatal intensive care units, molecular epidemiology data are scarce. Here, the authors describe two consecutive respiratory syncytial virus outbreaks caused by genotypes ON‐1 and NA‐2 in a neonatal intensive care unit in São Paulo, Brazil.
MethodsA prospective search for respiratory syncytial virus was performed after diagnosing the index case and four other symptomatic newborns in the neonatal intensive care unit. Nasopharyngeal aspirate samples of all patients in the neonatal intensive care unit were tested for 17 respiratory viruses using real‐time reverse transcriptase polymerase chain reaction. Genotyping was performed using nucleotide sequencing.
ResultsFrom May to August 2013, two different outbreaks were detected in the neonatal intensive care unit. A total of 20 infants were infected with respiratory syncytial virus‐A (ten and 14 with ON‐1 and NA‐2 genotypes, respectively). The mean age of the infants was 10 days, mean birth weight was 1,961g, and the mean gestational age was 33 weeks. Risk factors (heart disease, lung disease, and prematurity) were present in 80% and 85.7% of infants in the ON‐1 and NA‐2 groups, respectively. In total, 45.8% of infants were asymptomatic and 20.8% required mechanical ventilation. Coinfections were not detected during the outbreaks.
ConclusionsInfants in a neonatal intensive care unit who develop abrupt respiratory symptoms should be tested for respiratory viruses, especially respiratory syncytial virus. Even in the absence of severe symptoms, respiratory syncytial virus detection can prevent nosocomial transmission through infection control measures. A better understanding of respiratory syncytial virus molecular epidemiology is essential for developing new vaccines and antiviral drugs against respiratory syncytial virus.
O vírus sincicial respiratório é um patógeno frequentemente envolvido em surtos nosocomiais. Embora vários estudos tenham relatado tais surtos em unidades de terapia intensiva neonatal, os dados epidemiológicos moleculares são escassos. Neste artigo, descrevemos dois surtos consecutivos de vírus sincicial respiratório causados pelos genótipos ON‐1 e NA‐2 em uma unidade de terapia intensiva neonatal em São Paulo, Brasil.
MétodosUma busca prospectiva por vírus sincicial respiratório foi realizada após o diagnóstico do caso índice e outros quatro recém‐nascidos sintomáticos na unidade de terapia intensiva neonatal. Amostras de aspirado nasofaríngeo de todos os pacientes da unidade de terapia intensiva neonatal foram testadas para 17 vírus respiratórios com reação em cadeia da polimerase via transcriptase reversa em tempo real. A genotipagem realizada utilizando sequenciamento de nucleotídeos.
ResultadosDe maio a agosto de 2013, foram detectados dois surtos diferentes na unidade de terapia intensiva neonatal. Vinte e quatro crianças foram infectadas com vírus sincicial respiratório‐A (10 e 14 com os genótipos ON‐1 e NA‐2, respectivamente). A média da idade dos lactentes era de 10 dias, o peso médio ao nascer foi de 1961g e a idade gestacional média de 33 semanas. Fatores de risco (doença cardíaca, doença pulmonar e prematuridade) estavam presentes em 80% e 85,7% dos bebês nos grupos ON‐1 e NA‐2, respectivamente. No total, 45,8% dos lactentes eram assintomáticos e 20,8% necessitaram de ventilação mecânica. Não foram detectadas coinfecções durante os surtos.
ConclusõesBebês em unidade de terapia intensiva neonatal que desenvolvem sintomas respiratórios abruptos devem ser testados para vírus respiratórios, especialmente o vírus sincicial respiratório. Mesmo na ausência de sintomas graves, a detecção de vírus sincicial respiratório pode prevenir a transmissão nosocomial através de medidas de controle de infecção. Um melhor entendimento da epidemiologia molecular do vírus sincicial respiratório é essencial para o desenvolvimento de novas vacinas e drogas antivirais contra o vírus sincicial respiratório.
A infecção pelo vírus sincicial respiratório (VSR) é potencialmente fatal em lactentes, particularmente em bebês prematuros e com doença pulmonar crônica ou cardiopatia congênita. O VSR causa surtos graves em unidades de terapia intensiva neonatais (UTINs), leva à morbidade e mortalidade significativas em prematuros, além do aumento dos custos financeiros.1,2 Métodos convencionais de controle de infecção, como higiene das mãos e isolamento de pacientes por coortes, são recomendados; no entanto, esses procedimentos podem ser complementados pelo uso de palivizumabe (Synagis® ‐ MedImmune Inc., Gaithersburg, MD, EUA).2
O VSR é dividido nos tipos A (VSR‐A) e B (VSR‐B), com base em diferenças antigênicas derivadas de suas glicoproteínas G e F, e também categorizado em subtipos, com base no domínio variável da proteína G de ligação. O tipo A geralmente tende a ser mais dominante e está associado à maior gravidade da doença.3 A reinfecção pelo VSR é uma ocorrência comum, pois muitos genótipos podem circular concomitantemente. Essa variabilidade pode contribuir para a capacidade do vírus de causar surtos anuais e doenças graves.4,5 Além disso, relatos anteriores de surtos de infecção por VSR descreveram vários graus de gravidade da doença, mas não controlaram a presença de diferentes genótipos virais que podem apresentar virulências distintas.
No Brasil, há uma escassez de dados em neonatos em relação à epidemiologia molecular do VSR e o surgimento de novas cepas virais. Embora a circulação dos genótipos NA1 e ON1 do VSR já tenha sido descrita, o impacto sobre o curso da infecção por VSR em UTINs e possíveis surtos ainda precisa ser determinado.6–10 Relatamos dois surtos consecutivos de VSR causados por diferentes genótipos (ON1 e NA2) em uma UTIN brasileira. Como a detecção precoce de casos é crucial para a contenção da transmissão viral, objetivamos esclarecer a importância da detecção molecular e tipagem, assim como medidas de controle de infecção, nesse cenário desafiador.
Material e métodosCaracterísticas da UTINA Santa Casa de São Paulo é um hospital geral terciário no centro de São Paulo, com 2.000 nascimentos anuais em média. O Departamento de Obstetrícia é um serviço de referência em partos para programas municipais de gestação de alto risco e aproximadamente 15% dos recém‐nascidos necessitam de cuidados intensivos. A UTIN tem 28 leitos (14 na unidade de cuidados intermediários e 14 na unidade de terapia intensiva), divididos em quatro alas hospitalares fisicamente separadas (fig. 1). Os bebês na sala principal da UTI neonatal são mantidos em incubadoras fechadas ou em aquecedores radiantes abertos e muitos deles necessitam de ventilação mecânica (VM). Para bebês em incubadoras, a maioria dos cuidados de rotina são fornecidos através de portas laterais, mantidas fechadas quando não estão em uso. Aqueles na unidade de cuidados intermediários ficam em berços abertos ou incubadoras fechadas e não necessitam de VM.
Em média, mais de 65 profissionais de saúde têm contato físico diário com os bebês na UTIN: seis hospitalistas neonatais, oito estagiários neonatais, quatro residentes pediátricos, quatro estudantes de graduação em medicina, seis profissionais de enfermagem, 34 técnicos(as) de enfermagem e cinco terapeutas respiratórios. Após a higienização das mãos e a substituição das roupas pessoais por aventais (fornecidos pelo hospital), os pais têm permissão para visitar todas as alas e participar do cuidado de seus bebês. Crianças menores de 18 anos não podem entrar na UTIN.
Diagnóstico clínicoA infecção do trato respiratório superior foi definida quando tosse e rinorreia estavam presentes. O diagnóstico de bronquiolite foi feito com base nos seguintes sintomas: taquipneia, sibilo, tosse, crepitação e/ou abertura das narinas.
Investigação do surtoApós a identificação de um lactente sintomático com infecção confirmada por VSR, foram coletadas amostras de aspirado de nasofaringe (ANF) de todos os lactentes hospitalizados na UTIN. O controle do ANF foi repetido duas vezes por semana em todos os pacientes. As amostras foram enviadas ao Instituto de Ciências Biomédicas da Universidade de São Paulo.
O RNA viral foi extraído com o extrator automático NucliSens easyMAG (Biomerieux Inc., Durham, EUA), de acordo com as instruções do fabricante. A reação em cadeia de polimerase de transcrição reversa em tempo real (RT‐PCR, do inglês Real‐time reverse transcription polymerase chain reaction) foi feita com o kit AgPath‐ID One‐Step RRT‐PCR e com o 7300 Real‐Time PCR System (Applied Biosystems, Foster City, CA, EUA). Para identificar o patógeno responsável pelo surto, a RT‐PCR foi feita com o kit RRT‐PCR Panel Kit para vírus não influenza com o objetivo detectar 15 vírus respiratórios – grupo A e grupo B do VSR, metapneumovírus humano, adenovírus, coronavírus humano (HCoV‐HKU, ‐NL63, ‐OC43 e 229E), vírus parainfluenza humano (tipos 1, 2, 3 e 4), bocavírus humano, enterovírus humano e rinovírus humano, que foram fornecidos pelo CDC (Centro de Controle e Prevenção de Doenças, Atlanta GA, EUA),11 e primers e sondas comerciais para influenza A e B (Invitrogen, Carlsbad, CA, EUA).
As amostras positivas de qPCR foram amplificadas por PCR convencional two‐step para sequenciação de DNA: o cDNA foi sintetizado com o kit SuperScript III (Applied Biosystems, Foster City, EUA) de acordo com as instruções do fabricante. As análises da segunda região hipervariável do gene da proteína G foram feitas com PCR com os primers Gr5_fwd12 e FVAB_rev13 em uma mistura com 5μL de cDNA, 5μL de buffer de PCR [10X], [25mM] de cada: dNTP, [10pmol] de cada primer, 1,0U de Platinum® Taq DNA Polymerase (Invitrogen, Carlsbad, EUA) e água sem nuclease para um volume final de 25μL. A segunda etapa do semi‐nested PCR foi feita para amostras negativas da primeira PCR, com o primerforward GR5_fwd e o primer reverse F1AB_rev14 nas mesmas condições usadas para a PCR convencional. A amplificação foi feita no termociclador do tipo GeneAmp PCR System 9700® (Applied Biosystems, Foster City, EUA) com o seguinte programa: 95°C por 5 minutos, seguido de 35 ciclos, cada um com duração de 30 segundos a 95°C, 30 segundos a 55°C e 45 segundos a 72°C e, finalmente, cinco minutos de extensão a 72°C. Os produtos amplificados foram analisados por eletroforese em gel de agarose e visualizados sob luz ultravioleta após coloração com brometo de etídio. Os produtos amplificados do gene G de ∼1052 e 900 pb, produzidos por PCR convencional e semi‐nested PCR, consecutivamente, foram purificados com ExoSap‐IT (Affymetrix Inc., Santa Clara, EUA) e submetidos a um ciclo de reação de sequenciamento (Sanger) com um kit de coloração fluorescente com todos os primers, GR5, FV e F1AB, no aparelho 3100 DNA Sequencer (Applied Biosystems, Foster City, EUA). Ambas as fitas de cada produto de amplificação foram sequenciadas ao menos duas vezes. A edição das sequências, alinhamentos e análises filogenéticas foi feita com o software MegAlign 5.03v (DNAStar Inc., Madison, EUA). As sequências‐padrão publicadas do subgrupo A foram obtidas do GenBank (https://www.ncbi.nlm.nih.gov/genbank) como referência de diferentes linhagens e genótipos.
ÉticaO estudo foi aprovado pelo comitê local de ética em pesquisa.
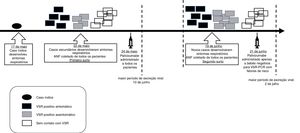
ResultadosPrimeiro surto (maio)O caso‐índice envolveu um bebê do sexo masculino prematuro de 30 dias (33 semanas de idade gestacional) com um peso ao nascer de 2.290g, hospitalizado desde o nascimento devido à prematuridade e à displasia broncopulmonar. Estava em desmame da suplementação de oxigênio na unidade de tratamento intermediário quando começou a apresentar tosse, coriza e febre. A insuficiência respiratória desenvolveu‐se rapidamente, exigiu VM. Cinco dias depois, cinco bebês (dos 17 que estavam na UTIN) desenvolveram sintomas respiratórios; portanto, um surto foi declarado (fig. 2). No mesmo dia, amostras de ANF foram coletadas de todos os pacientes na UTIN para a detecção do vírus respiratório por RT‐PCR. Dez bebês estavam infectados com o VSR, dos quais apenas três compartilhavam a ala com o caso índice. A taxa de ataque foi de 58,8% e apenas uma fatalidade ocorreu (Taxa de Letalidade=10%). No entanto, a morte não foi atribuída à infecção pelo VSR, uma vez que o paciente não apresentava sintomas respiratórios e tinha hipoplasia pulmonar congênita, considerada a causa do óbito. Informações adicionais são dadas na tabela 1.
Características clínicas dos pacientes infectados pelo VSR no primeiro surto (causado pelo genótipo ON‐1) e segundo surto (causado pelo genótipo NA‐2)
| 1 surto: VSR‐tipo A ON‐1 (n=10) | 2° surto: VSR‐tipo A NA‐2 (n=14) | |
|---|---|---|
| Características | ||
| Idade gestacional (média, semanas) | 34,9 | 33,4 |
| Peso ao nascer (média, gramas) | 2327 | 1700 |
| Idade no início (média, dias) | 35,4 | 14,3 |
| Comorbidades | ||
| Doença cardíaca congênita | 2 (20%) | 1 (7,1%) |
| Doença pulmonar | 1 (10%) | ‐ |
| Prematuridade (< 37 semanas) | 5 (50%) | 11 (78,6%) |
| Sepse concomitante ou anterior de início tardio | 3 (30%) | 5 (35,7%) |
| Apresentação clínica e desfechos | ||
| Assintomático | 3 (30%) | 8 (57,1%) |
| Infecção respiratória superior leve | 2 (20%) | 1 (7,1%) |
| Bronquiolite sem VM | 4 (40%) | 1 (7,1%) |
| Bronquiolite com VM | 1 (10%) | 4 (28,6%) |
| Mortes | 1 (10%) | 2 (14,3%) |
Infecção do trato respiratório superior: presença de tosse, rinorreia; bronquiolite: taquipneia, sibilo, tosse, crepitação e/ou abertura nasal; VM, ventilação mecânica.
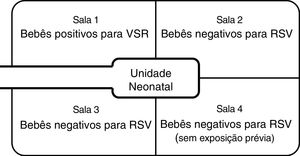
Medidas de precauções de contato e respiratórias foram imediatamente implantadas. Todos os bebês da UTIN foram separados em três coortes diferentes. Lactentes VSR‐positivos foram colocados na enfermaria 1 sob precauções de contato e respiratórias. Os lactentes negativos para o VSR que tiveram contato prévio com qualquer um dos bebês positivos para o VSR foram colocados nas alas 2 e 3 apenas com precauções de contato. Lactentes negativos para o VSR que nunca tiveram contato com os pacientes positivos para o VSR foram colocados na ala 4.
A equipe de saúde também foi dividida entre as alas e sua programação de rotação interna foi suspensa. Cinquenta integrantes da equipe tiveram amostras de ANF e saliva testadas para vírus respiratórios, com resultado negativo em todas. As visitas à UTIN foram limitadas e sua duração reduzida. As medidas de controle de infecção foram reforçadas para a equipe de saúde e para as famílias dos bebês pelo grupo de controle de infecção e membros da UTIN e incluíram higiene das mãos, limpeza ambiental e aumento da conscientização sobre sintomas respiratórios. No segundo dia do surto, o fármaco palivizumabe foi administrado a todos os bebês na UTIN.
O controle do ANF (teste para os 17 vírus respiratórios, como descrito anteriormente) foi feito duas vezes por semana em todos os bebês na UTIN, de modo que a duração da excreção viral e possíveis coinfecções pudessem ser determinadas. O acompanhamento continuou por quatro semanas após a última amostra positiva ter sido identificada. Os bebês VSR‐positivos foram mantidos isolados até a alta da UTIN, foram testados semanalmente por PCR até que um resultado negativo fosse alcançado. O período mais longo de excreção viral foi de 29 dias.
Segundo surto (junho)Durante os 24 dias seguintes, nenhum outro lactente na UTIN desenvolveu sintomas sugestivos de infecção por VSR; portanto, o primeiro surto foi considerado controlado. Trinta e quatro dias após o caso índice, um novo surto foi identificado e as medidas de controle foram restabelecidas (fig. 2). Quatorze lactentes apresentaram VSR, identificado nas secreções respiratórias (tabela 1). O palivizumabe foi administrado apenas a bebês com resultado negativo na PCR para VSR com fatores de risco (oito pacientes). Neste segundo surto, a taxa de ataque foi de 63,6% e duas fatalidades ocorreram (Taxa de Letalidade=14,3%). Ambos os bebês eram prematuros e estavam sob VM quando a infecção pelo VSR foi detectada. Suas mortes foram atribuídas à sepse bacteriana; no entanto, o VSR pode ter contribuído. O tempo de excreção viral mais longo foi de 14 dias.
Características dos bebês infectadosEntre os 24 bebês positivos para o VSR, 15 (62,5%) nasceram prematuramente com menos de 37 semanas de gestação (tabela 1). A média de idade gestacional foi de 33 semanas (28±40 semanas), a média de idade ao diagnóstico foi de 27,5 dias (variação de um a 80 dias) e a média de peso ao nascer foi de 1961g (variação de 765 a 3.440g). Onze lactentes (45,8%) eram assintomáticos. Entre os bebês sintomáticos, 12,6% apresentavam sintomas respiratórios superiores leves (tosse ou coriza) ou febre, 20,8% apresentavam bronquiolite sem necessidade de VM e 20,8% necessitaram de suporte ventilatório invasivo. Seis lactentes eram elegíveis pelas diretrizes brasileiras para receber palivizumabe.15 Em ambos os surtos, os dados disponíveis não foram suficientes para estabelecer fatores de risco independentes para infecção por VSR.
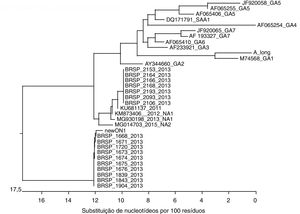
Caracterização genotípicaA análise molecular foi feita para todas as 24 amostras positivas para o VSR (fig. 3). Os vírus isolados durante o primeiro surto foram caracterizados como VSR‐tipo‐A ON‐1, enquanto todas as infecções durante o segundo surto foram causadas pelo VSR‐tipo‐A NA‐2. A análise de aminoácidos evidenciou uma assinatura genética característica nas amostras do segundo surto. Nenhuma coinfecção viral foi detectada em nenhum dos surtos.
DiscussãoQue seja de nosso conhecimento, este é o primeiro relato de dois surtos neonatais consecutivos causados por dois genótipos distintos de VSR no Brasil. Em conjunto, os episódios envolveram 24 lactentes em uma UTIN de alta complexidade e ocorreram no período pós‐pico do VSR (as maiores taxas de circulação viral ocorrem entre março e maio em São Paulo). O ensaio de RT‐PCR e a análise da sequência genética foram cruciais na confirmação dos dois surtos distintos e na adoção de medidas de controle, incluindo o uso do palivizumabe.
Os bebês nascidos prematuramente e com doença cardíaca ou pulmonar subjacente são mais propensos a desenvolver sintomas graves quando infectados com o VSR. Em comparação com infecções adquiridas na comunidade, as infecções nosocomiais estão associadas a uma mortalidade significativamente maior.2 As taxas de ataque nos surtos de VSR em UTIN relatadas na literatura variam de 9 a 55%.16–21 Nossas taxas de ataque foram muito maiores, chegaram a 58,8% durante o primeiro surto e, inesperadamente, 63,6% no segundo surto. Tal disparidade pode ser atribuída ao fato de que a maioria dos nossos pacientes era presumivelmente susceptível aos novos genótipos descritos e nenhum deles havia recebido palivizumabe antes do início do surto. A ausência de imunidade protetora nos bebês e em suas mães pode ter permitido a reprodução e a transmissão mais eficientes do vírus. Além disso, esses genótipos emergentes parecem ter uma vantagem de aptidão em relação aos genótipos anteriores do mesmo grupo de VSR.22
Em contrapartida, não observamos um aumento na frequência de doença grave, porque apenas 10% dos pacientes no grupo ON‐1 e 28,6% no grupo NA‐2 desenvolveram insuficiência respiratória e necessitaram de VM. Tal frequência pode ser explicada pela idade gestacional média relativamente alta (34 semanas) de nossos pacientes. Embora não tivéssemos bebês prematuros extremos, todos os cinco bebês infectados por VSR que necessitaram de VM nasceram prematuramente com menos de 33 semanas, o que reforçou a contribuição da prematuridade como um importante fator de risco para infecção grave por VSR.
Não há diretrizes para o uso de palivizumabe durante surtos em instituições de saúde. Identificamos pequenos estudos que usaram o palivizumabe em UTINs como um componente dos procedimentos de controle de surtos.17–20 Quando o primeiro surto foi detectado em nosso meio, todos os 17 pacientes da UTIN receberam palivizumabe, enquanto o palivizumabe estava disponível apenas para pacientes negativos para o VSR no segundo surto, devido à quantidade limitada de medicação disponível e seu alto custo. Embora a minoria dos bebês tivesse uma indicação rigorosa para receber o palivizumabe, acreditamos que a administração ampla e imediata do medicamento possa ter contribuído para o controle oportuno e a baixa morbidade/mortalidade na população estudada.
Estudos anteriores descrevem a cocirculação dos grupos A e B do VSR em períodos epidêmicos, inclusive em populações brasileiras.5,7,9,23,24 O VSR‐A foi responsável por ambos os surtos; no entanto, nossos casos ocorreram em uma unidade fechada. Contudo, identificamos dois genótipos diferentes de VSR‐A na UTIN: ON‐1 e NA‐2. O primeiro é um novo genótipo com grandes duplicações de aminoácidos na glicoproteína G de ligação. Foi relatado pela primeira vez em dezembro de 2010 em Ontário, no Canadá, e tem se espalhado rapidamente por todo o mundo.22,25–30
A ocorrência do VSR e genótipos específicos durante surtos nosocomiais pode refletir a circulação do vírus na comunidade. Isso é parcialmente demonstrado pelo tempo de nossos surtos, que respeitam a sazonalidade em nossa região.
Em resumo, o diagnóstico etiológico precoce é especialmente relevante para terapia antiviral e medidas de isolamento para prevenir surtos hospitalares, reduz a necessidade de procedimentos diagnósticos adicionais e o tempo de internação hospitalar. Um melhor entendimento da epidemiologia molecular do VSR é essencial para o desenvolvimento de vacinas e medicamentos antivirais e outras medidas de controle contra a infecção por VSR.
Conflitos de interesseOs autores declaram não haver conflitos de interesse.
Como citar este artigo: Silva DG, Almeida FJ, Arnoni MV, Sáfadi MA, Mimica MJ, Jarovsky D, et al. First report of two consecutive respiratory syncytial virus outbreaks by the novel genotypes ON‐1 and NA‐2 in a neonatal intensive care unit. J Pediatr (Rio J). 2020;96:233–9.